-
[+Code]imbalanced한 데이터를 처리하는 sampling기법(over, under, SMOTE)Machine Learning/Feature 2020. 10. 21. 22:31
현실에 있는 많은 데이터들은 불균형합니다. 예를 들어 제조업에서 제품의 불량을 분류하기 위해 10,000개의 row를 가진 데이터셋을 구축하였다고 하면, 우리가 원하는 label은 대부분이 '정상'으로 나올 것입니다. 왜냐하면 대부분의 공정에서 불량품이 나오지 않기 때문이죠. 10,000개의 데이터셋에서 9900개의 데이터가 '정상'으로 나온다면 이 데이터셋으로 제대로 된 모델링을 할 수 없습니다. 이런 imbalanced한 데이터를 oversampling, undersampling, SMOTE의 데이터 resampling 기법을 통해 어느정도 해소할 수 있습니다. 먼저 아래의 데이터셋을 확인해보도록 하겠습니다.
참조 : www.kaggle.com/rafjaa/resampling-strategies-for-imbalanced-datasets
Resampling strategies for imbalanced datasets
Explore and run machine learning code with Kaggle Notebooks | Using data from Porto Seguro’s Safe Driver Prediction
www.kaggle.com
Imbalanced Data
흔히 볼 수 있는 이진분류의 label입니다. 0이 굉장히 많고 1이 적습니다. 이 상태로 학습을 하게 되면 모든 데이터를 0이라고 예측하더라도 정확도는 90%가 넘게 나올 것입니다. 별로 의미가 없는 정확도죠.

Oversampling vs Undersampling

Original dataset은 위와 같이 굉장히 imbalanced한 데이터입니다.
Undersampling이란 minority class(주황)의 크기만큼 majority class(파랑)를 sampling하는 기법입니다. 이 기법의 가장 큰 단점은 information loss가 굉장히 크다는 것입니다.
Oversampling이란 반대로 majority class의 크기만큼 minority class를 copy하는 기법입니다. 이 기법의 가장 큰 단점은 oversample되지 않은 외부의 데이터에 굉장히 민감하게 반응할 수 있다는 것입니다. 즉, overfitting의 가능성이 굉장히 커지게 됩니다.
Python Code
python에서는 imblearn이라는 라이브러리를 통해 sampling을 진행할 수 있습니다.
import imblearn from sklearn.datasets import make_classification X, y = make_classification( n_classes=2, class_sep=1.5, weights=[0.9, 0.1], n_informative=3, n_redundant=1, flip_y=0, n_features=20, n_clusters_per_class=1, n_samples=100, random_state=10 ) df = pd.DataFrame(X) df['target'] = y df.target.value_counts().plot(kind='bar', title='Count (target)');이진분류의 label을 90%와 10%로 설정하여 가상의 데이터셋을 만들었습니다.
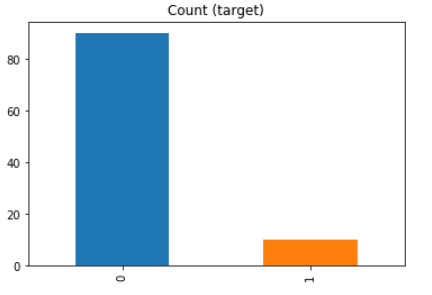
위와 같이 label이 설정되어 있는 데이터셋을 가지고 여러가지 sampling을 진행하겠습니다. 그리고 생성한 데이터를plot_2d_space함수를 통해 확인합니다.
# 데이터셋을 scatter plot으로 보기 위한 함수 def plot_2d_space(X, y, label='Classes'): colors = ['#1F77B4', '#FF7F0E'] markers = ['o', 's'] for l, c, m in zip(np.unique(y), colors, markers): plt.scatter( X[y==l, 0], X[y==l, 1], c=c, label=l, marker=m ) plt.title(label) plt.legend(loc='upper right') plt.show()plotting을 위해 pca를 사용해서 2차원으로 차원축소합니다. 앞으로 sampling에 이 데이터를 사용합니다.
from sklearn.decomposition import PCA pca = PCA(n_components=2) X = pca.fit_transform(X) plot_2d_space(X, y, 'Imbalanced dataset (2 PCA components)')
Random under-sampling and over-sampling
1. RandomUnderSampler
Random으로 minority class에 맞춰 majority class를 sampling합니다. minority class(주황)가 10개이므로 majority class도 10개 sampling됩니다.
from imblearn.under_sampling import RandomUnderSampler rus = RandomUnderSampler() X_rus, y_rus = rus.fit_sample(X, y) id_rus = rus.sample_indices_ print('sampled indices:', id_rus) plot_2d_space(X_rus, y_rus, 'Random under-sampling')
10%의 비율인 class=1에 맞춰 class=0이 줄어든 모습
2. RandomOverSampler
위와는 반대로 majority class(파랑)에 맞춰 minority class(파랑)를 duplicate합니다.
from imblearn.over_sampling import RandomOverSampler ros = RandomOverSampler() X_ros, y_ros = ros.fit_sample(X, y) print(X_ros.shape[0] - X.shape[0], 'new random picked points') plot_2d_space(X_ros, y_ros, 'Random over-sampling')
class=1인 instance의 개수가 바뀌지 않은 것처럼 보이지만 실제로는 80개의 instance가 추가되었고 겹쳐서 하나처럼 보이는 것입니다. 확인해보면 class=0의 개수는 90개 classs=1의 개수도 90개입니다.

Under-sampling : Cluster Centroids
Cluster method를 바탕으로 한 Under-sampling 기법입니다. sampling_strategy의 딕셔너리값에 따라 전체 데이터셋에서 몇 개의 중심을 만들어내는지를 정하여 데이터를 sampling합니다. 가령 {0:30}이라면 전체 majority class의 instance에서 30개의 중심값을 sampling하는 것이죠.
from imblearn.under_sampling import ClusterCentroids cc = ClusterCentroids(sampling_strategy={0:30}) X_cc, y_cc = cc.fit_sample(X, y) plot_2d_space(X_cc, y_cc, 'Cluster Centroids under-sampling')
{0:30}인 경우 instance 
{0:10}인 경우 instance Under-Sampling : Tomek links
Tomek links는, 매우 가까운 instances들의 쌍을 나타냅니다. 두 개의 class사이에 공간을 확보하기 위해 인접해있는 majority class를 지우는 방법입니다. 변수들 사이의 hyperplane을 생성하는 SVC에서 좀 더 효율적으로 작동하지 않을까하는 개인적인 생각입니다.

from imblearn.under_sampling import TomekLinks tl = TomekLinks( sampling_strategy='majority') X_tl, y_tl = tl.fit_sample(X, y) id_tl = tl.sample_indices_ print('Removed indexes:', len(X)-len(id_tl)) plot_2d_space(X_tl, y_tl, 'Tomek links under-sampling')
지워진 instance는 4개밖에 되지 않아 잘 표시가 나지는 않지만 기존 dataset과 비교하여 4개의 majority class가 줄었습니다. Tomek links의 쌍을 이루고 있는 majority class의 instance가 4개 지워진 것입니다.
Over-Sampling : SMOTE
SMOTE란, (Synthetic Minority Oversampling TEchnique)의 줄임말이며, minority class에 있는 elements들의 사이를 k-nearest neighborhood의 방법으로 보간하는 기법입니다. 아래의 그림처럼 minority class에 있는 elements사이를 채우는 기법이라고 할 수 있습니다.
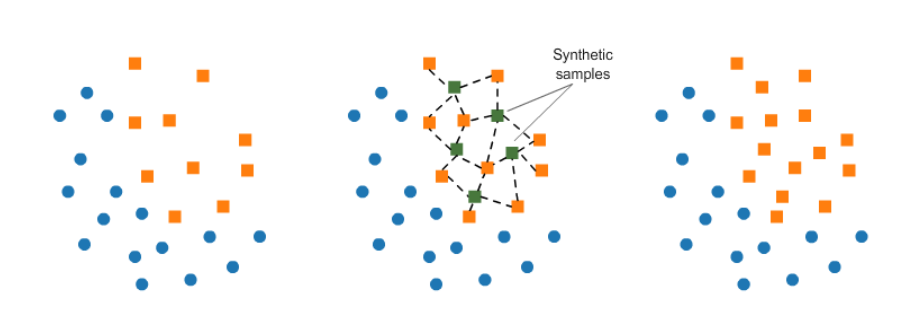
from imblearn.over_sampling import SMOTE smote = SMOTE(sampling_strategy="minority", random_state=1000) X_sm, y_sm = smote.fit_sample(X, y) print("만들어진 instances : ", len(X_sm)-len(X)) plot_2d_space(X_sm, y_sm, 'SMOTE over-sampling')
SMOTE 기법을 통해 minority class 인 1의 instance가 80개 증가하였다는 것을 확인할 수 있습니다.
Over-Sampling followed by under-sampling
참조한 홈페이지에서 Over-Sampling과 under-Sampling을 혼합한 방법을 제시하였습니다. 그것은, SMOTE와 Tomek links기법을 combination하는 것입니다.
from imblearn.combine import SMOTETomek smt = SMOTETomek(sampling_strategy='auto', random_state=1000) X_smt, y_smt = smt.fit_sample(X, y) print((len(X_smt))-len(X)) plot_2d_space(X_smt, y_smt, 'SMOTE + Tomek links')
SMOTE 기법으로 oversampling을 한 후, Tomek links 기법을 사용하여 class = 0 과 class = 1이 인접해있는 majority class를 지우는 방법입니다. 총 74개의 minority class(주황)를 생성하였습니다.
사실 이 방법이 imbalanced한 데이터셋에 effective하다고 소개를 하고 있는데, 캐글을 찾아보다 어떤 분이 남긴 코멘트가 흥미로웠습니다. '이런 oversampling 등의 방법이 데이터의 imbalanced를 대처하는 효과적이라면 더 주목을 받아야하는 것 아닌가요? 혹시 이 기법 효과를 가지는 것이 제한적이라거나... 그런 이유는 아닌가요?'라는 코멘트였는데 저도 사실 어느 정도 공감이 됩니다.
실제로 이진분류에서 model의 performance를 향상시키기 위해 처음 접한 기법인데 기법을 적용했을 때도 performance가 향상되지 않았기 때문에 의구심을 가지던 찰나라서 좀 더 와닿았던 것 같습니다. 언제 효과적으로 적용시킬 수 있을지 조금 더 알아봐야 하겠습니다 ...
'Machine Learning > Feature' 카테고리의 다른 글
Feature Encoding 관련 Article (0) 2021.03.25 Time Series 데이터를 변수로 Encoding하는 다양한 기법 (0) 2021.03.02